De dianas a dsRNA: un nuevo enfoque computacional que impulsa el desarrollo de biofungicidas basados en RNAi
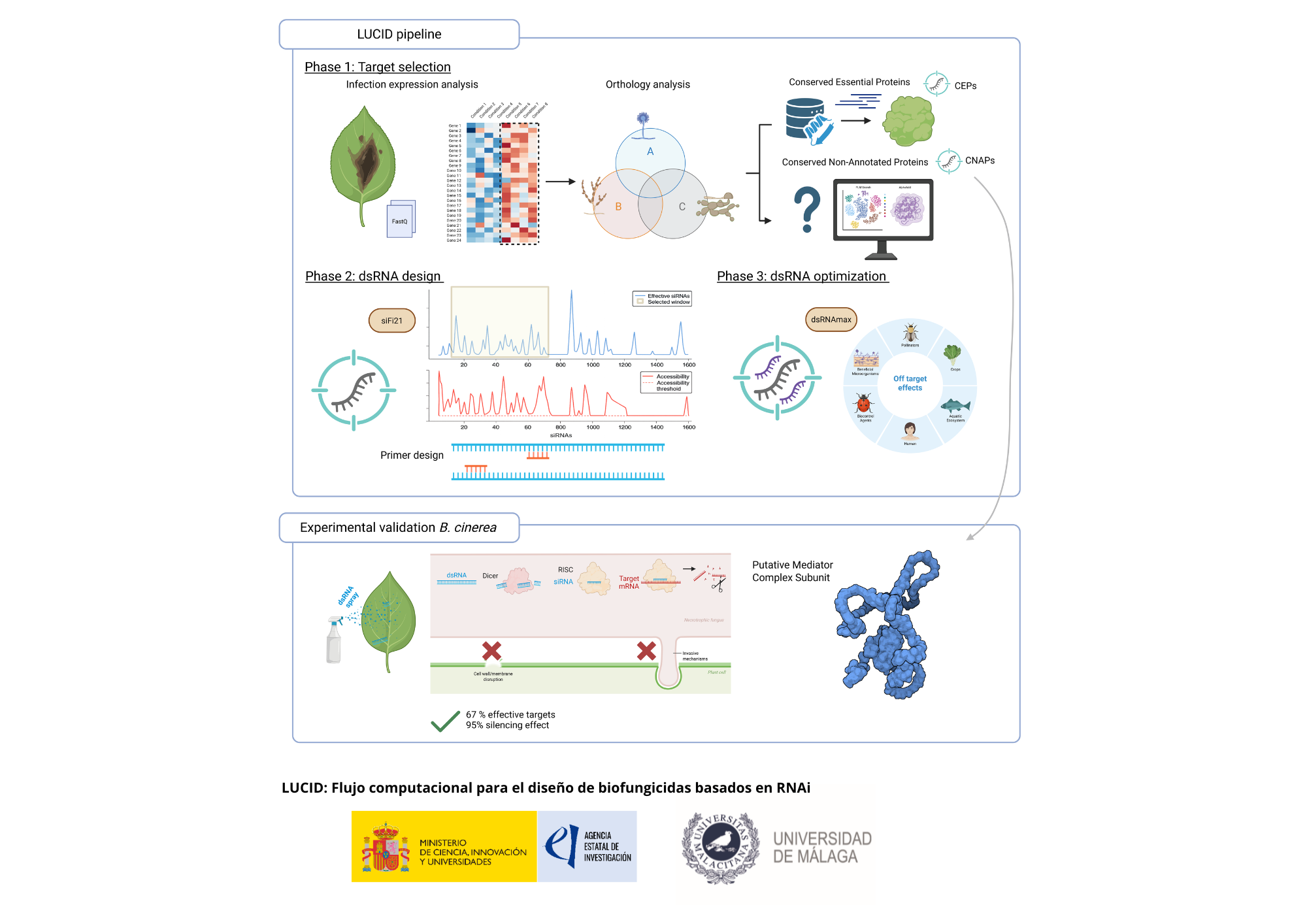
Las enfermedades causadas por hongos representan una amenaza creciente para la producción agrícola, mientras que la aparición de resistencias y las limitaciones ambientales reducen la eficacia de muchos fungicidas convencionales. En un nuevo artículo de la revista Plant Biotechnology Journal, investigadores del IHSM La Mayora (UMA–CSIC) presentan LUCID (Locating Uncovered, conserved, and Indispensable for pathogenicity Determinants), un pipeline computacional que acelera el desarrollo de biofungicidas basados en dsRNA (ARN de doble cadena), una alternativa más específica y potencialmente más sostenible que actúa silenciando genes del patógeno mediante interferencia de ARN. LUCID integra tres pasos clave: identificación de dianas, diseño del dsRNA y predicción de posibles efectos fuera de objetivo. Para localizar los mejores genes a silenciar, aplica una estrategia doble que detecta tanto (i) proteínas esenciales conservadas (CEPs) como (ii) proteínas conservadas no anotadas (CNAPs), combinando datos transcriptómicos con genómica comparada en múltiples especies fúngicas. La validación en Botrytis cinerea (moho gris) mostró una alta eficacia: el 67% de las dianas propuestas se silenciaron con éxito, con una eficiencia media de silenciamiento del 96%. Además, al combinar LUCID con modelos de lenguaje de proteínas, se identificó un nuevo determinante de patogenicidad en B. cinerea: una posible proteína del complejo mediador. En el artículo participaron los investigadores del IHSM La Mayora Lucía Jiménez-Castro, Alba López-Laguna, Dolores Fernández-Ortuño, Alejandro Pérez-García y Álvaro Polonio (autor de correspondencia)
Ir a la noticia